Nature Genetics IF=30.8|非编码seRNA m6A修饰介导组蛋白修饰和癌基因表达
发布时间:2024-04-03 15:21 | 点击次数:
2023年11月13日,中山大学肿瘤防治中心研究者在Nature Genetics发表了题为 Super-enhancer RNA m6A promotes local chromatin accessibility and oncogene transcription in pancreatic ductal adenocarcinoma 的研究成果。通过m6A MeRIP-seq、PAR-CLIP-seq,RNA-seq等测序技术发现首次揭示了胰腺癌中seRNA m6A修饰对组蛋白修饰和癌基因表达的调控,扩展了人们对超级增强子及其转录本功能新的认知。

标题: Super-enhancer RNA m6A promotes local chromatin accessibility and oncogene transcription in pancreatic ductal adenocarcinoma
发表时间:2023/11/13
发表期刊:Nature Genetics
影响因子:IF=30.8/Q1
研究方法:m6A meRIP-seq、RNA-seq、PAR-CLIP-seq、CoIP-MS、RNA pull down、CUT&Tag-seq、ATAC-seq
研究摘要
目前,人们对于非编码RNA m6A修饰的生物学功能仍知之甚少。本研究描绘了胰腺导管腺癌( PDAC )中超级增强子RNA ( seRNA )的 m6A修饰情况,并揭示了m6A seRNA、H3K4me3修饰、染色质可及性和癌基因转录的调控轴。
研究证明了在PDAC中过表达的cofilin家族蛋白CFL1作为METTL3的辅助因子,有助于seRNA m6A甲基化的形成。增加的se RNA m6A修饰被reader YTHDC2识别,YTHDC2招募H3K4甲基转移酶MLL1,促进H3K4me3共转录修饰。具有高水平H3K4me3的超级增强子增加染色质的可及性并促进癌基因的转录。总的来说,这些结果揭示了CFL1 - METTL3-seRNA m6A-YTHDC2/MLL1轴在局部染色质状态和基因表达的表观遗传调控中发挥作用,这加强了对超级增强子及其转录本功能的认识。
技术路线

研究结果
(1)PDAC seRNAm6A修饰图谱
通过ENCODE和GEO数据库中正常胰腺和PDAC的H3K27ac ChIP-seq 数据,共注释到18311个超级增强子区域,在65个PDAC和33个正常组织的RNA-seq中也检测到了上述seRNA,其中17467个seRNA在10%以上样本中均表达(图a)。超级增强子标志物H3K27ac,H3K4me3和H3K4me1在识别到的区域中心上下游2kb范围内富集,而阴性对照H3K27me3没有富集。
对65个PDAC ( 33例配对样本,32例非配对样本)中的seRNA进行m6A meRIP-seq ,在17467个seRNA中检测到5375个(30.77 % )带有m6A修饰(图a),且分布在整个seRNA中(图b ),富含m6A典型RRACH基序(图c)。其中共有410个差异m6A seRNA,其中398个m6A seRNA (97.07 %)在PDAC中上调,12个下调(2.93%)(图d),IGV可视化与m6A -meRIP-qPCR结果均表明多个seRNA m6A水平在PDAC中高于正常组织,有16个seRNAs m6A上调与PDAC患者较短的无进展生存( PFS )时间显著相关(HR >1.00; (FDR) P < 0.05)。
(2)CFL1在PDAC中seRNA m6A的异常形成中起关键作用
为了探究PDAC seRNA m6A异常增加的原因,研究人员在数据库中搜索了与m6A常见writer(METTL3、METTL14、WTAP和K97%AA1429)相互作用的RNA结合蛋白(RBPs),发现CFL1蛋白对seRNA m6A水平贡献分数最高(图e),且显著相关(图f)。通过RNA-seq以及数据库数据表明CFL1 mRNA水平在PDAC中显著高于正常组织(图g ),在广州和北京采集的另外2个样品中也验证了这些结果(图h),对30对配对的PDAC和正常样本的进行WB,PDAC的CFL1蛋白水平也明显高于正常组织。KM生存曲线和多因素Cox比例风险模型分析显示,高CFL1水平(>=中位数)的PDAC比低CFL1水平( <中位数)的具有更短的生存时间。

(3)CFL1是seRNA m6A形成过程中的METTL3辅助因子
对PANC-1细胞进行CFL1敲除和NC进行了m6A meRIP-seq,结果显示有3217(77.07%)个seRNAs m6A水平下调,占在PDAC中m6A上调的seRNA 69.10 % (图b )。通过CFL1 PAR-CLIP-seq发现大多数CFL1结合峰位于远端基因间和内含子远端,且与CFL1-KO组下调m6A区域大幅重叠。因CFL1可能通过ROCK / LIMK / cofilin通路促进癌症进展,因此研究人员通过m6A meRIP-seq检测在Cyto D处理及NC中m6A修饰,Cyto D处理并没有对seRNA m6A整体修饰水平产生影响,说明PDAC细胞中CFL1产生的seRNA m6A可能不依赖于ROCK / LIMK / cofilin通路,而是依赖于核内CFL1本身。
CoIP实验及RNA Pull down-WB证明了CFL1和METTL3的直接相互作用(图d),免疫荧光实验显示CFL1与METTL3在PANC-1细胞核中存在共定位(图e),结构域检测显示METTL3锌指结构域( ZnF1 ,氨基酸259 ~ 298)是CFL1结合所必需的(图f)。
通过METTL3 KO vs NC m6A meRIP-seq检测了METTL3对总RNA和核RNA中seRNA m6A水平的影响,发现METTL3- KO细胞中seRNA m6A降低与CFL1-KO的下调的seRNA大部分相同(图h)。PAR-CLIP-seq分析还表明,CFL1和METTL3在CFL1 KO相关的m6A下调 seRNAs的TSS附近富集。RIP-qPCR验证了CFL1或METTL3与这些seRNA存在相互作用,并且在敲除细胞中,m6A水平显著降低。CFL1-KO细胞中seRNAs(n =2373,图2k)的METTL3结合信号显著降低,RIP - qPCR证实了这一点(图2l )。进一步利用dPspCas13b - CFL1系统在CFL1 - KO细胞中验证了CFL1的功能,表明CFL1是seRNA m6A修饰的METTL3辅助因子。

(4)SeRNA m6A通过YTHDC2 - MLL1相互作用使H3K4me3甲基化
m6A通常由reader介导发挥作用,结合已发表数据,发现m6A seRNA上的YTHDC2结合密度较高,YTHDC2 PAR-CLIP-seq结果显示YTHDC2在基因间区和内含子区富集(图f ),且结合位点几乎与m6A seRNAs重叠,RNA Pull down-WB验证了YTHDC2与m6A se RNA的相互作用。RIP-qPCR分析表明,CFL1 -或METTL3 - KO导致YTHDC2显著降低。通过dPspCas13b - METTL3系统在CFL1 - KO细胞中验证了m6A、YTHDC2和H3K4me3的作用,YTHDFC2突变实验完全抑制了YTHDC2对m6A - seRNAs的识别。YTHDC2 - KO与CFL1-KO一样同样降低了H3K4me3峰。
YTHDC2 CoIP -MS鉴定到MLL1富集,CoIP-WB验证了YTHDC2与MLL1的相互作用,免疫荧光实验显示YTHDC2与MLL1在细胞内存在共定位。YTHDC2,MLL1和H3K4me3共定位于m6A seRNA相关的染色质上,并且MLL1和H3K4me3信号在YTHDC2-KO上减弱(图d )。CUT&Tag-seq和核RNA PAR-CLIP-seq结果显示,YTHDC2在超级增强子中的定位与seRNA中的YTHDC2有很好的重叠,还发现了YTHDC2、MLL1和H3K4me3共定位,并且YTHDC2 - KO显著降低MLL1或H3K4me3信号(附图3a)。这些结果表明,m6A seRNAs通过YTHDC2和MLL1相互作用,在转录水平上共同促进了局部染色质H3K4me3修饰。

(5)m6A seRNAs引起染色质开放和致癌基因转录
为了研究CFL1-m6A-YTHDC2/MLL1-H3K4me3轴对染色质可及性的影响,在CFL1-KO、YTHDC2-KO或MLL1 - KO或non - KO PANC - 1细胞中通过ATAC-seq分析了可及性染色质,当CFL1,YTHDC2或MLL1被敲除时,有10300个共有区域ATAC缺失,主要富集在内含子、远端基因间区和启动子区域(图a,b )。
RNA-seq结果显示,与non- KO相比,1743个基因在CFL1 - KO中显著下调,富集于缺氧和mTORC1信号传导(图d ,右)等11个癌症相关过程,这些基因在数据集及PDAC数据库中均显著上调。此外结合数据集,发现图d所示的通路得分与CFL1-YTHDC2 - MLL1特征得分或肿瘤样本和单细胞样本中的或CFL1水平之间存在显著相关性(图f)。高水平的MICAL2和AMIGO2 (由两个hyper-m6A seRNA调控)与患者较短的PFS显著相关。这些结果表明,m6A se RNA相关的CFL1 - YTHDC2 / MLL1轴在重塑染色质可及性和癌基因转录中发挥重要作用。

(6)m6A seRNA相关的CFL1促进PDAC细胞的恶性表型
最后研究了CFL1是否对PDAC细胞的恶性表型有影响,发现异位过表达CFL1显著促进PDAC细胞在体外和体内的增殖、迁移、侵袭和转移,而CFL1 - KO具有相反的作用,同时还发现METTL3 -KO或YTHDC2 - KO显著抑制PDAC细胞恶性表型。将野生型METTL3或缺失CFL1结合区域的突变型METTL3转染到CFL1过表达和METTL3沉默的细胞中,结果显示野生型METTL3恢复了恶性细胞表型,而突变型METTL3恢复了恶性细胞表型。综上所述,这些结果表明m6A seRNA形成相关的CFL1在PDAC中发挥致癌作用。
云序生物m6A修饰研究五大模块
01 m6A RNA修饰测序
GLORI(Glyoxal and nitrite-mediated deamination of unmethylated adenosine)技术是针对m6A修饰开发的单碱基检测技术。实现了真正意义的高效率、高灵敏度、高特异性、无偏好单碱基m6A位点检测,并对m6A位点的修饰水平进行绝对定量。
对m6A RNA甲基化,目前流行的检测手段为m6A-MeRIP-Seq技术,适用于m6A RNA甲基化谱研究,快速筛选m6A RNA甲基化靶基因。云序可提供mRNA和多种非编码RNA的m6A测序:
m6A 全转录组测序(涵盖mRNA,LncRNA,circRNA)
m6A LncRNA测序(涵盖LncRNA和mRNA)
m6A Pri-miRNA测序(涵盖Pri-miRNA和mRNA)
m6A mRNA测序
m6A miRNA测序
02检测整体m6A修饰水平
比色法检测整体RNA修饰水平
快速检测m6A整体甲基化水平
03 m6A RNA修饰上游酶的筛选
m6A RNA修饰相关酶PCR芯片
寻找上游直接调控m6A RNA甲基化的甲基转移酶。
04 m6A RNA修饰靶基因验证
MeRIP-qPCR/GenSeq® MeRIP试剂盒
云序提供各类不同修饰的MeRIP-qPCR服务以及销售GenSeq® MeRIP试剂盒,可针对mRNA,lncRNA,环状RNA等不同类型的RNA分子进行检测,低通量验证RNA修饰靶基因表达水平。
05机制互作研究
5.1 RIP-seq/qPCR/GenSeq® RIP试剂盒
筛选或验证RNA修饰直接靶点,研究RNA修饰靶基因的调控机制。
5.2 RNA pull down -MS/WB
筛选或验证目标RNA互作基因或蛋白,研究相应的分子调控机制。
5.3ChIP-seq
筛选或验证目标蛋白与DNA互作,研究相应的分子调控机制。
5.4Ribo-seq
筛选翻译水平发生变化的RNA分子,研究相应的分子调控机制。
5.5SLAM-seq
筛选稳定性发生变化的RNA分子,研究相应的分子调控机制。
5.6CHIRP-seq
筛选或验证目标RNA与DNA互作,研究相应的分子机制。
云序生物RNA修饰多组学研究相关产品
m6A RNA甲基化测序
m5C RNA甲基化测序
m1A RNA甲基化测序
m7G RNA甲基化测序
ac4C RNA乙酰化测序
O8G RNA氧化修饰测序
2’-O-RNA甲基化测序
m6Am RNA甲基化测序
BID-seq假尿嘧啶测序
RNA pulldown
RNA-seq
RIP测序
Ribo-seq
SLAM-seq
ATAC-seq
CUT&Tag-seq
云序生物服务优势
优势一:云序累计支持客户发表数百篇SCI论文,合计影响因子3000 +
优势二:累计完成测序样本数万例,全面覆盖医口、农口等各类样本。
优势三:全面检测mRNA和各类非编码RNA(circRNA,lncRNA,Pri-miRNA等)。
优势四:提供RNA修饰一站式服务:整体水平检测、RNA修饰研究、MeRIP-qPCR验证、RIP-seq、RNA pull-down、Ribo-seq和SLAM-seq等。
优势五:率先研发超微量MeRIP测序技术,RNA量低至500ng起。
优势六:国内较全的RNA修饰测序平台,提供m6A、m5C、m1A、m7G、m3C、O8G、ac4C乙酰化和2'-O-甲基化、假尿嘧啶等修饰的测序技术。
云序客户RNA修饰部分文章列表
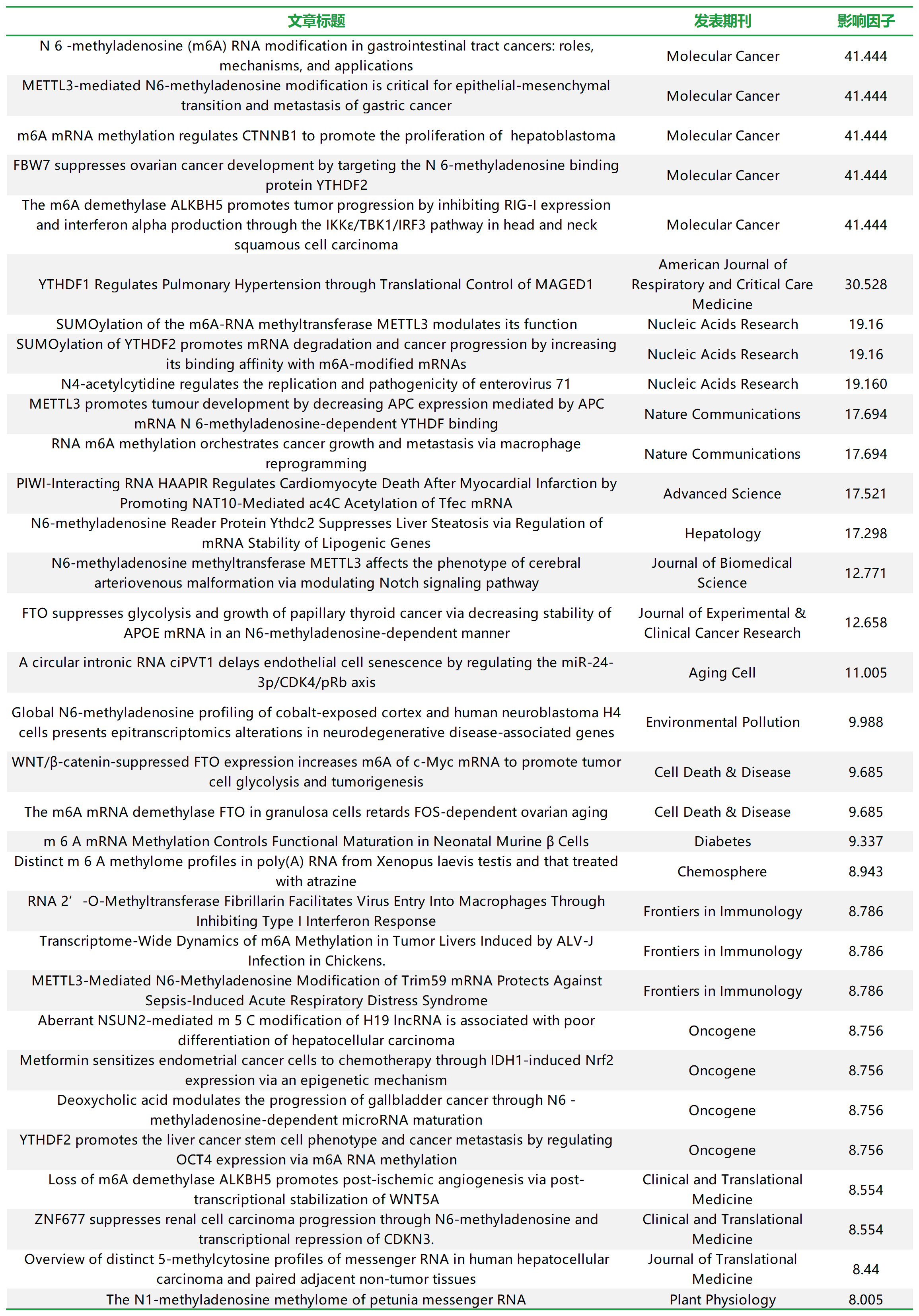
往期回顾
用户文章IF=15.1|余健秀团队再次携手云序,揭示TBK1介导的AGO2磷酸化促进肺癌发生
云序客户余健秀课题组m6A方向再次取得重大发现——SUMO化促进YTHDF2结合m6A修饰的mRNA影响癌症进展
1区 IF: 21|云序MeRIP-Seq & RIP-Seq助力肺动脉高压中m6A阅读蛋白YTHDF1机制研究
RIP-seq项目文章|nature子刊揭示低氧诱导AGO2线性泛素化调控肿瘤发生发展机制
云序客户m6A高分文章|揭示组蛋白乙酰化与m6A修饰在眼部黑色素瘤发生中的共同作用机制
用户文章 IF=13.6|AHR介导的 m6A RNA甲基化是 PM2.5 诱导斑马鱼幼体心脏畸形的原因之一
用户文章 IF=19.2|范先群院士团队利用m1A MeRIP-seq联合多组学测序揭示眼黑色素瘤调控新机制
云序客户| m6A MeRIP-seq助力揭示早发糖尿病表观调控新机制
客户文章|IF=10.6—甲状腺癌中m5C甲基转移酶NSUN2通过维持tRNA的稳定性促进密码子依赖性的致癌翻译
云序客户| MeRIP-seq+RIP-seq技术揭示 m6A 甲基化调控BMSCs成骨分化新机制
云序客户m6A高分文章|揭示组蛋白乙酰化与m6A修饰在眼部黑色素瘤发生中的共同作用机制
用户文章m6A专题|IF=9.8|m6A去甲基化酶ALKBH5缺乏会加重钴致神经退行性损伤
客户文章| Nature子刊 揭示了FMR1通过m6A修饰调控早期胚胎发育的分子机制
用户文章IF=14.9 1区:ac4C乙酰化调节人胚胎干细胞的自我更新
云序助力解析新城疫病毒感染的鸡巨噬细胞的m6A表达谱
用户文章 1区 lncRNA m6A甲基化测序助力人脂肪干细胞成骨分化的调控机制研究
云序用户农口IF20+论文:植物mRNA存在ac4C新型修饰,对RNA稳定性及翻译产生重要影响




