包学包会 | 单碱基分辨率类测序项目的 IGV 可视化查看
发布时间:2023-02-02 10:22 | 点击次数:
Itegrative Genomics Viewer (IGV)是由Broad研究院的研究人员开发的一款功能强大的综合性基因组学可视化工具,借助于此工具,从事生物信息的科研工作者就可以轻松的分析查看多种基因组数据。除了直观的查看序列信息及遗传密码中的序列变化或突变外,还可以查看拷贝数变化、染色质沉淀数据和表观遗传学修饰等。
云序生物课堂曾经介绍过基于免疫沉淀技术类型的项目(包括 MeRIP 测序、MeDIP 测序、ChIP 测序、ATAC 测序等)如何使用 IGV 软件对抗体富集峰进行可视化展示。然而,有一些特殊类型的 RNA 或 DNA 修饰测序项目,因为实验原理上的特殊性,使得在 IGV 软件当中打开 .bam 文件后,看到的图像很不一样。
下面,我们就为大家介绍一下 m5C/5mC(Bis-seq)、2‘-O-甲基化修饰(Nm-seq)、m7G/m3C 修饰(AlkAniline-seq)这三种单碱基分辨率类项目的 IGV 可视化结果的查看方法。
软件安装以及需要用到的文件
首先,进入IGV的官方下载地址 ,选择适合您电脑的版本进行下载和安装。在云序生物的报告文件压缩包当中,也包含了一个 Windows 版本的 IGV 软件,您也可以直接解压运行。**运行可能需要一段时间初始化IGV,请您耐心等待。
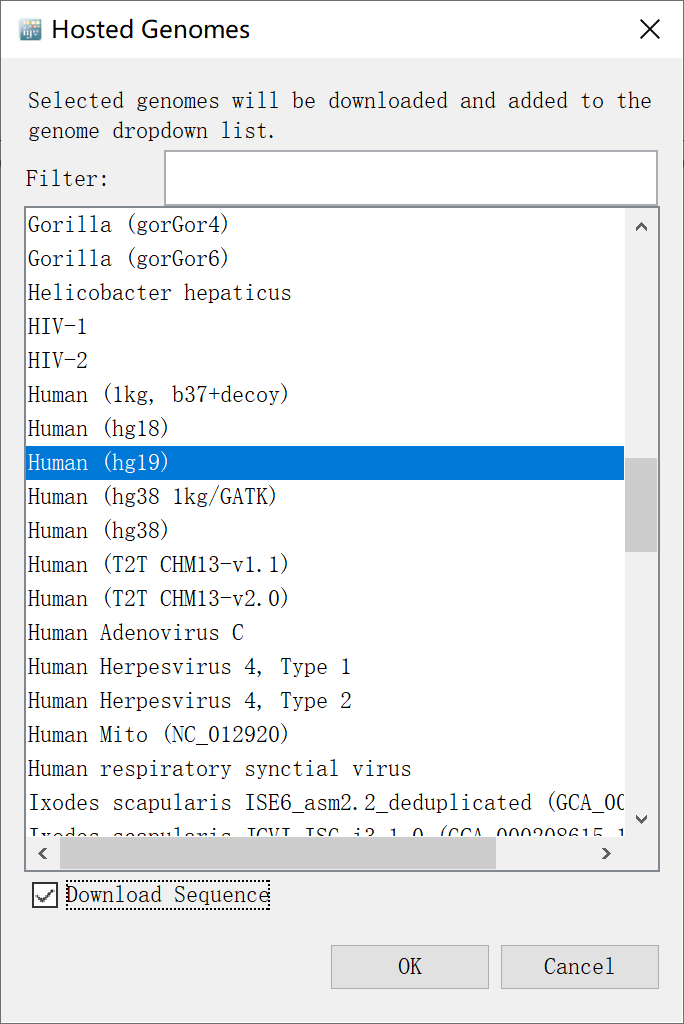
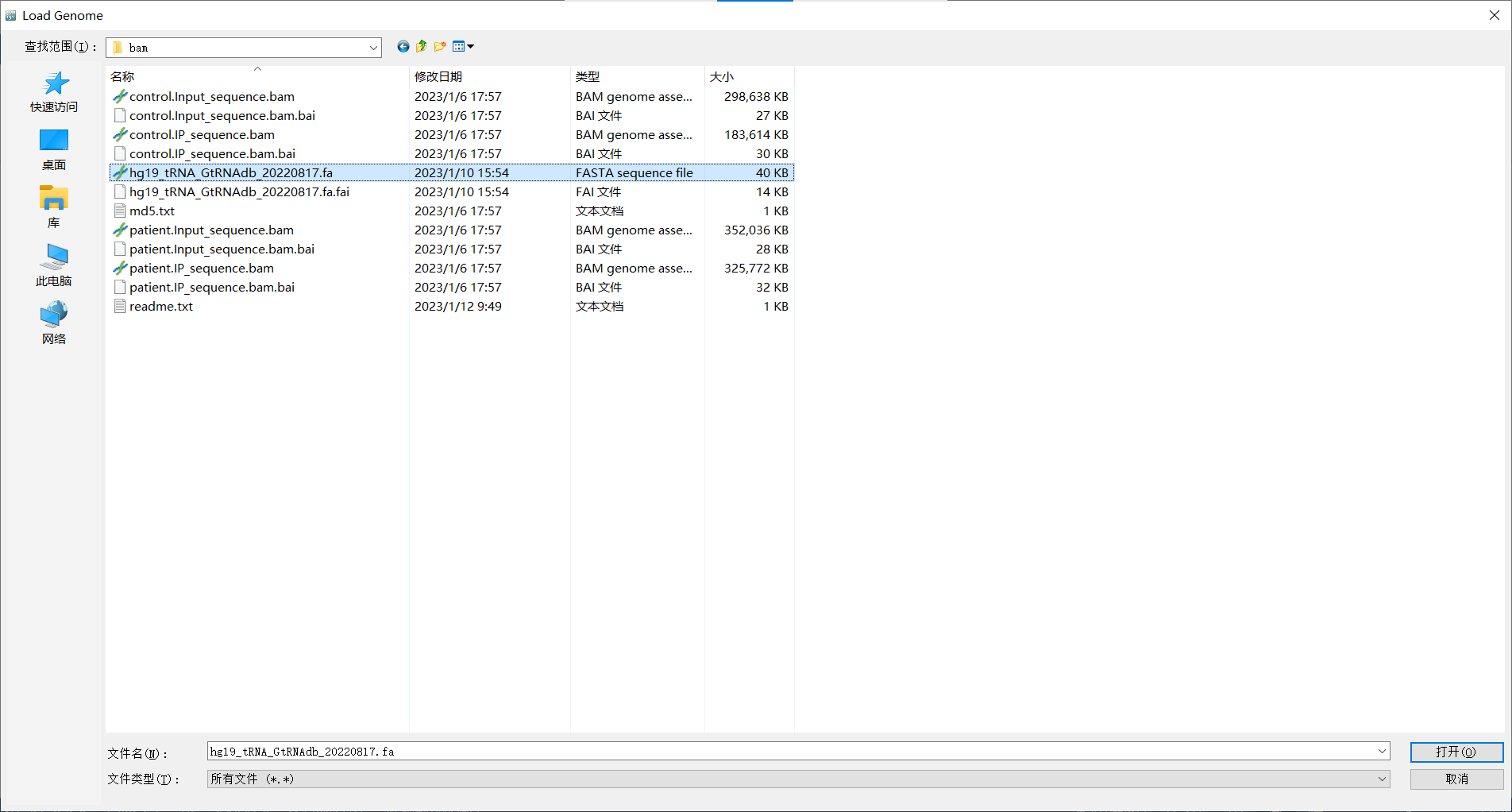
完成安装并进入 IGV 软件后,首先我们需要加载参考基因组。对于常见物种,您可以直接使用 IGV 官方自带的参考基因组(例如下方截图当中所示的人类基因组版本 hg19),操作如下:选择菜单-Genomes-Select Hosted Genome。建议您勾选“Download Sequence“,以将基因组下载到本地电脑,方便以后使用。
m5C/5mC 修饰(Bis-seq)
RNA 的 m5C Bis-seq 以及DNA 的 5mC WGBS,其原理都是利用了重亚硫酸盐(Bisulfite)这种化学试剂的特性,可以将未经修饰的胞嘧啶(C)转变为尿嘧啶(U),进而在后续的测序当中被读取为胸腺嘧啶(T);而倘若胞嘧啶上发生了 5 号位上的甲基化修饰(m5C/5mC),则重亚硫酸盐将无法造成碱基改变,进而在后续的测序当中仍被读取为胞嘧啶(C)。除了重亚硫酸盐法以外,DNA 的 EM-seq 也使用一系列酶起到了类似的选择性碱基转化的作用。总之,对于这一类 m5C/5mC 单碱基精度测序实验而言,发生了 m5C/5mC 修饰的位点将在测序结果当中以一定的比例呈现为 C。
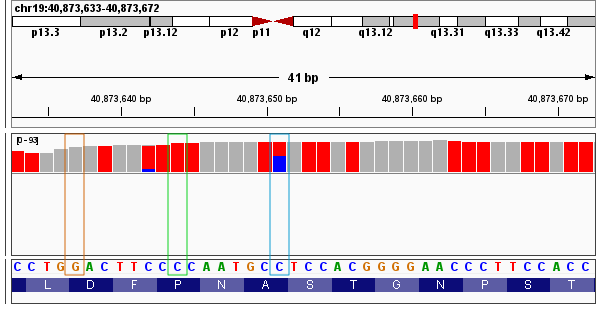
对于大部分参考基因组上的 A、T、G位点,表示其覆盖度的柱状图都是灰色的,表示测序结果与参考基因组上的碱基类型完全一致。例如图中橙色方框所框住的位点,其参考基因组在此处是 G,而实际测序结果在此处也是 G。
对于大部分参考基因组上的 C 位点,表示其覆盖度的柱状图是红色的,这表示在该位点上实际测序测得的结果是 T。例如图中绿色方框所框住的位点,其参考基因组在此处是 C,但因为重亚硫酸盐的转化,实际测序结果在此处则是 T。
对于少部分参考基因组上的 C 位点,表示其覆盖度的柱状图是部分红色+部分蓝色的,这表示在该位点上实际测序测得的结果是部分 T + 部分 C,说明该位点上存在一定比例的 m5C 甲基化修饰现象。例如图中蓝色方框所框住的位点,其参考基因组在此处是 C,但因为该位点上实际存在一定比例的 m5C 修饰现象,加之重亚硫酸盐的转化,实际测序结果在此处则是 33 次 T(红色)和 38 次 C(蓝色)。
2‘-O-甲基化修饰(Nm-seq)
RNA 核糖上 2 号位上的羟基如果被氧甲基(-O-CH3)取代,就会形成2‘-O-甲基化修饰,又称 Nm 修饰。在特殊化学试剂条件下,氧化-消除-脱磷酸循环(OED 循环)可以从 RNA 的 3’ 端逐个剪下单个核苷酸(1 nt);而如果 RNA 上的第 N 位存在 2‘-O-甲基化修饰,则 OED 循环反应将会有很大概率在第 N 位的 3’ 端停止下来,从而在后续的测序当中读取到大量 3’ 端断裂于第 N 位的测序 reads,呈现“断崖状”的特征。
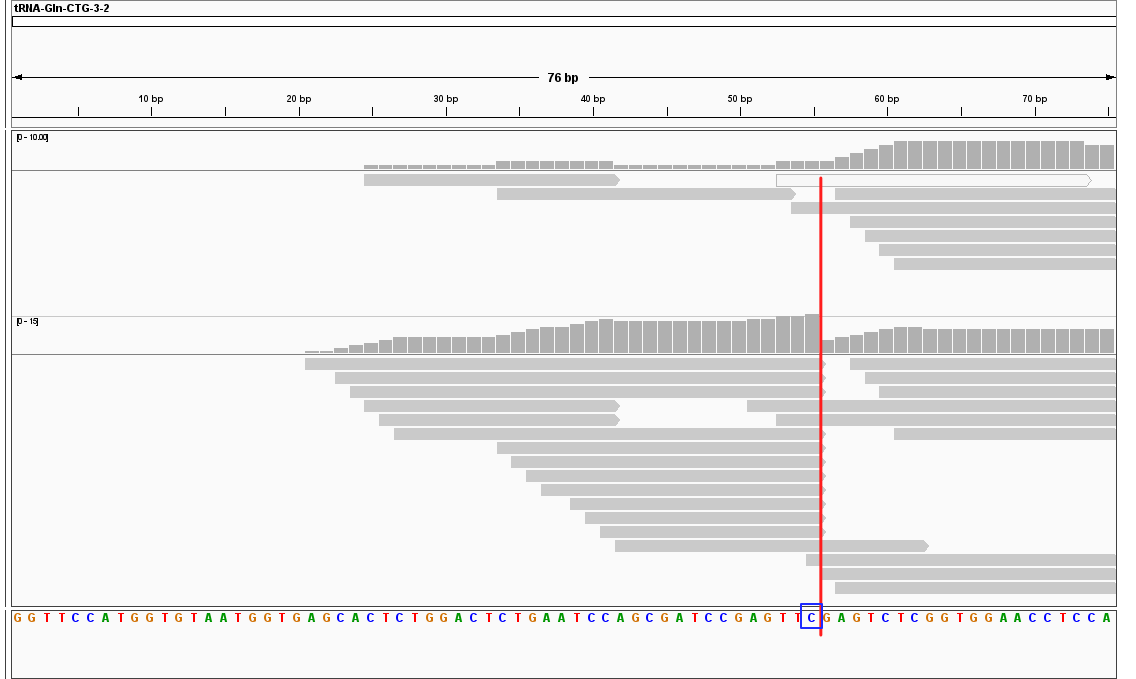
我们可以看到,在上方例图展示的区域范围之内,Input 样品的测序 reads 分布比较随机,并没有在某一位点出现集中的现象;而在 OED 样品中,则在蓝色方框所表示的第 55 位 C 的 3’ 端出现了红色竖线所示的“断崖式” reads 集中的现象——如果我们进行数量统计,会发现蓝色方框中的 C 位点,存在 15 个测序 reads,其中多达 11 个测序 reads 的 3‘ 端就“断”在此处,比例高达 73.33%,彰显第 55 位 C 存在很大概率发生了 2‘-O-甲基化修饰。
m7G/m3C 修饰(AlkAniline-seq)
RNA 上的 m7G 或 m3C 修饰,可以通过一系列特殊的化学处理来探明,这种测序方法简称为 AlkAniline-seq。在一系列特殊化学试剂条件下,m7G 或 m3C首先会发生脱碱基反应,如果 RNA 上的第 N 位存在 m7G 或 m3C 修饰,随后有很大概率在第 N+1 位的 5’ 端形成 RNA 链的断裂,从而在后续的测序当中读取到大量 5’ 端断裂于第 N+1 位的测序 reads,呈现“断崖状”的特征。
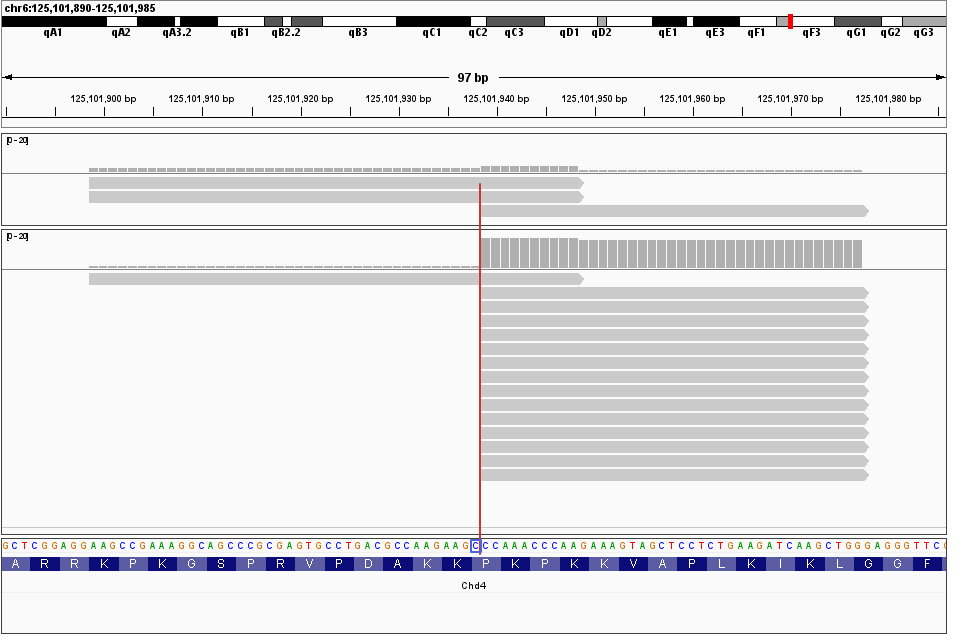
例如上面这幅例图当中,展现了小鼠 mm10 参考基因组上 chr6: 125101938 位 C 的 m3C修饰情况。该图包含了同一个样品的两种不同处理方法后的测序结果:上方是 Input 样品,即未经特殊化学试剂处理,直接建库测序的结果;下方是 AlkAniline 化学试剂处理样品进行建库测序的结果。对于每一种处理类型的样品,柱状图表示该位点处的覆盖度(Coverage),由于它们都与参考基因组完美匹配(没有任何突变现象),因此都显示为灰色的方柱;片段图则表示了每一个测序 reads 的位置以及方向(用箭头表示)。
我们可以看到,在上方例图展示的区域范围之内,Input 样品的测序 reads 分布比较随机,并没有在某一位点出现集中的现象;而在 AlkAniline 处理的样品中,则在蓝色方框所表示的chr6: 125101938 位 C的“N+1”位,也就 chr6: 125101939 位的 5’ 端出现了红色竖线所示的“断崖式” reads 集中的现象——如果我们进行数量统计,会发现chr6: 125101939 位上存在 15 个测序 reads,其中多达 14 个测序 reads 的 5‘ 端就“断”在此处,比例高达 93.33%,彰显其上游 chr6: 125101938 位用蓝色方框所表示的 C 存在很大概率发生了 m3C 修饰。
相 关 产 品
往期回顾
超详细图文版:如何实现m6A测序数据可视化?
GEO 数据上传指南
技术干货:SRA数据上传指南
5 分钟时间解锁环状 RNA 序列查询技能
技术干货|MeRIP-qPCR方法及应用细谈

地址: 松江区莘砖公路518号18号2楼
网址: http://www.cloud-seq.com.cn
电话: 021-64878766
邮箱: market@cloud-seq.com.cn




