1 区,IF = 19.16| 云序 RNA 乙酰化测序 acRIP-seq 助力病毒复制机制研究!
发布时间:2022-10-18 15:13 | 点击次数:
N4-乙酰胞苷 (ac4C) 对 mRNA 的翻译和稳定性至关重要。虽然在 RNA 病毒中发现了 ac4C,但 ac4C 影响病毒复制的详细机制尚不清楚。2022 年 8 月 16 日,云序客户中国科学院武汉病毒所与深圳大学的研究者们在 Nucleic Acids Research(IF = 19.16)杂志上发表了文章「N4-acetylcytidine regulates the replication and pathogenicity of enterovirus 71」。本文报道了肠道病毒 71(EV71) 基因组的 5』UTR 区存在由宿主乙酰转移酶 NAT10 催化产生的 ac4C 修饰。而且,ac4C 通过选择性招募 PCBP2 到 IRES 来增强病毒 RNA 的翻译,并提高了 RNA 的稳定性。此外,抑制 NAT10 和突变 ac4C 位点抑制了 EV71 的复制,且 ac4c 缺陷突变体 EV71 在小鼠体内表现出的致病性降低。此项研究结果突出说明了 ac4C 在 EV71 感染中的重要作用,并为潜在的抗病毒治疗提供了新的见解。云序生物有幸参与了此项研究中的 ac4C acRIP-seq 高通量技术服务。
至此我司已经为合作客户发在 Nucleic Acids Research(IF = 19.16)、Journal of Hematology & Oncology(IF = 23.168)、Advanced Science 杂志(IF = 17.52)等核心期刊中均提供过 ac4C acRIP-seq 高通量技术服务以及生信分析。

发表期刊:Nucleic Acids Research
影响因子:19.160
发表时间:2022年8月16日
研究方法:ac4C acRIP-seq、acRIP-qPCR、RIP-qPCR
文章链接:N4-acetylcytidine regulates the replication and pathogenicity of enterovirus 71

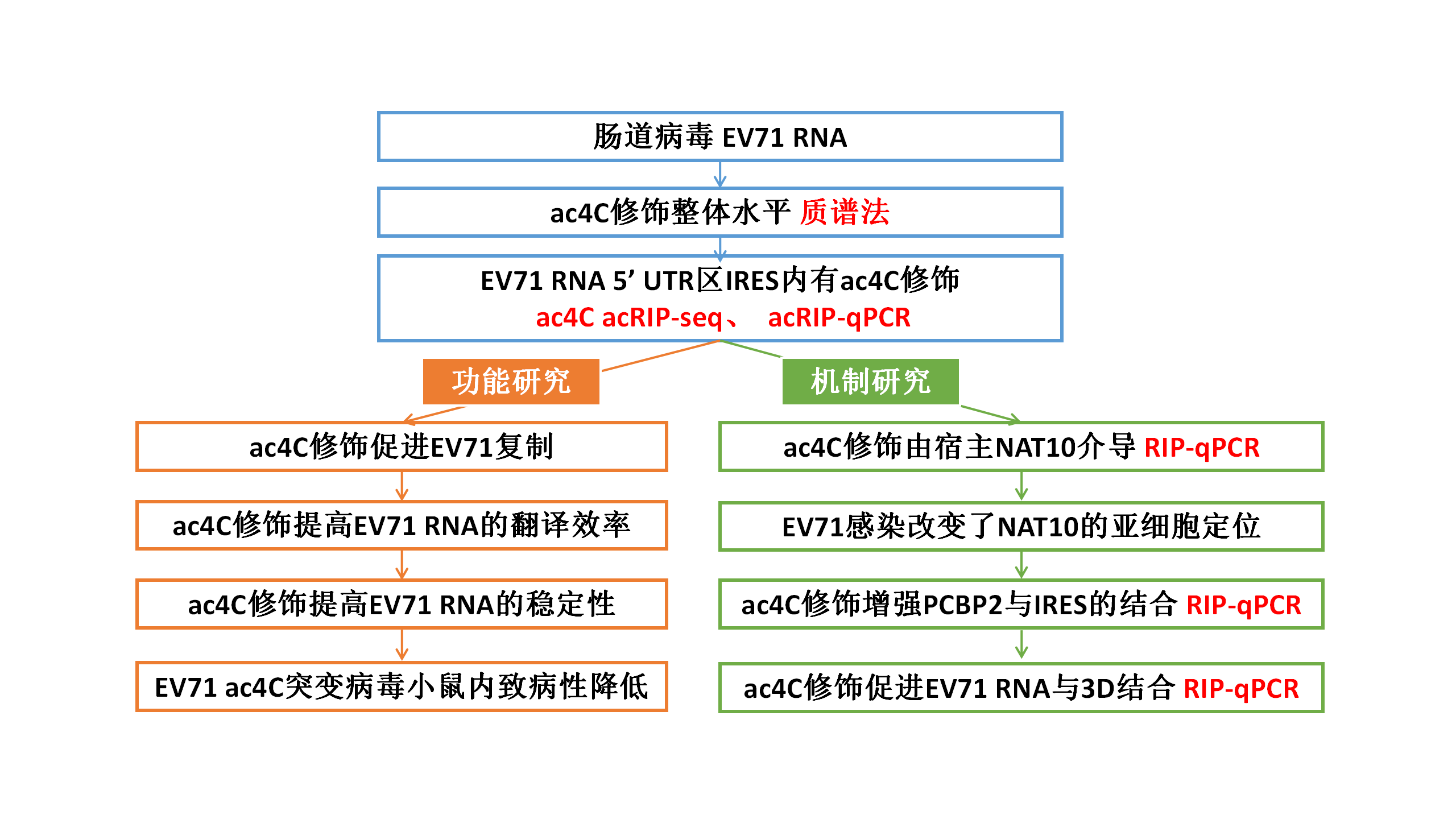
01 EV71 RNA 的 5’ UTR 中含有 ac4C 修饰,且宿主 NAT10 表达模式发生改变
为了研究 EV71 RNA 是否含有 ac4C 修饰,作者大规模纯化 EV71 RNA,并通过 UPLC-MS/MS 进行定量,发现病毒粒子的 EV71 RNA 中 ac4C 残基占总胞苷的 0.201%。此外,针对 EV71 基因组 RNA 进行 acRIP-qPCR 分析,发现来自病毒感染细胞和病毒粒子的 EV71 RNA 都被拉下,而在体外转录的病毒基因组没有被拉下。为了绘制 EV71 基因组中的 ac4C 修饰谱,作者进行了 ac4C acRIP-seq 分析。在 EV71 基因组的 311 到 406 核苷酸 (nt) 区域发现了一个明显的 ac4C 峰,该峰位于 5』 UTR 区 IRES 内的茎环 IV(nt 242-445)。EV71 不编码任何具有乙酰转移酶活性的蛋白。为了研究 EV71 RNA 上的 ac4C 修饰是否被细胞乙酰转移酶 NAT10 催化,作者进行了 RIP-qPCR 实验,结果表明 NAT10 与 EV71 RNA 相互作用。此外,过表达或敲低 NAT10 时,病毒 RNA 上的 ac4C 的强度相应增加或降低。综上所述,EV71 RNA 上含有 ac4C 修饰,且由宿主乙酰转移酶 NAT10 介导。EV71 在细胞质中进行复制,而 NAT10 主要分布在细胞核中,而免疫荧光显示,在 EV71 存在的情况下,NAT10 同时定位于细胞核和细胞质,表明 EV71 感染改变了 NAT10 的亚细胞定位。此外,在病毒感染过程中检测到较强的 NAT10 信号,提示 EV71 感染可能增强了 NAT10 的表达。
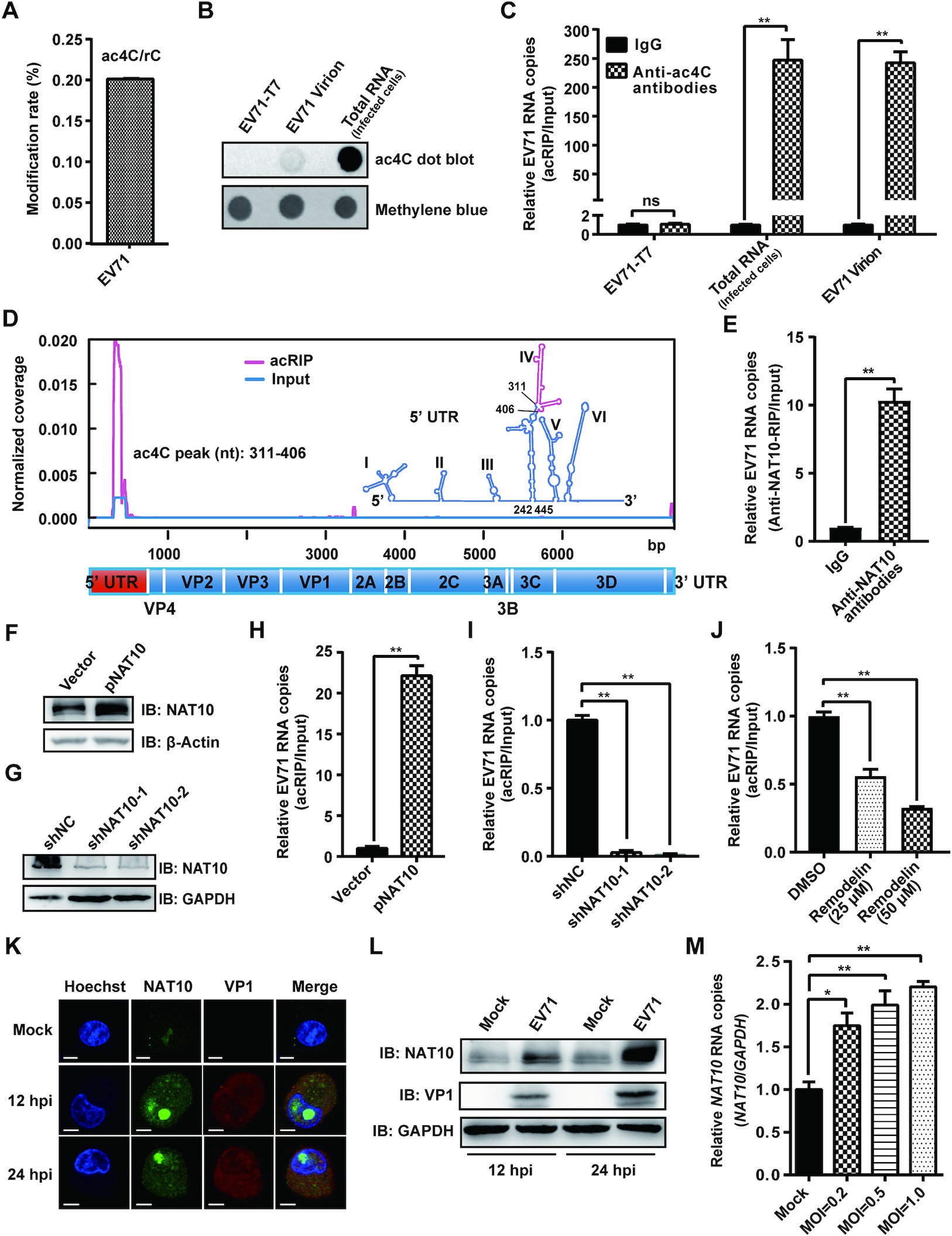
图 1. EV71 含有 ac4C 修饰,并改变了 NAT10 的表达模式
02 5’ UTR 区的 ac4C 修饰促进了 EV71的复制
为了研究 ac4C 是否影响 EV71 的复制,作者构建了 NAT10 敲低 Vero 细胞并感染 EV71,发现 EV71 滴度和 RNA 拷贝数均显著降低,暗示 NAT10 促进了病毒的复制。NAT10 作用于共识基序 5』-CCG-3』,促进中心胞苷的乙酰化。作者为了评估 EV71 5』 UTR 上的 ac4C 峰中的两个 5』-CCG-3』基序是否被 ac4C 修饰,构建了 EV71 cDNA 全长克隆的突变体 Mut-331、Mut-350、Mut-331-350 以及对照 Mut-383。acRIP-qPCR 结果显示,与 WT EV71 相比,ac4C 突变病毒粒子和突变病毒感染细胞的 EV71 RNA 中的 ac4C 丰度均显著降低,表明这些 C 残基在 EV71 基因组中发生乙酰化。且当 ac4C 位点发生突变时,子代病毒滴度和基因组 RNA 拷贝数均显著下降,而在 shRNA 介导的 NAT10 敲除的细胞中,WT 和突变病毒的子代病毒滴度没有差异。这些结果支持了 ac4C 在 EV71 复制中起着重要作用。在 EV71 复制过程中存在正链和负链 RNA,与负链 RNA 相比,下调 NAT10 更显著地降低了病毒正链 RNA 的水平,表明 NAT10 主要影响病毒正链 RNA。
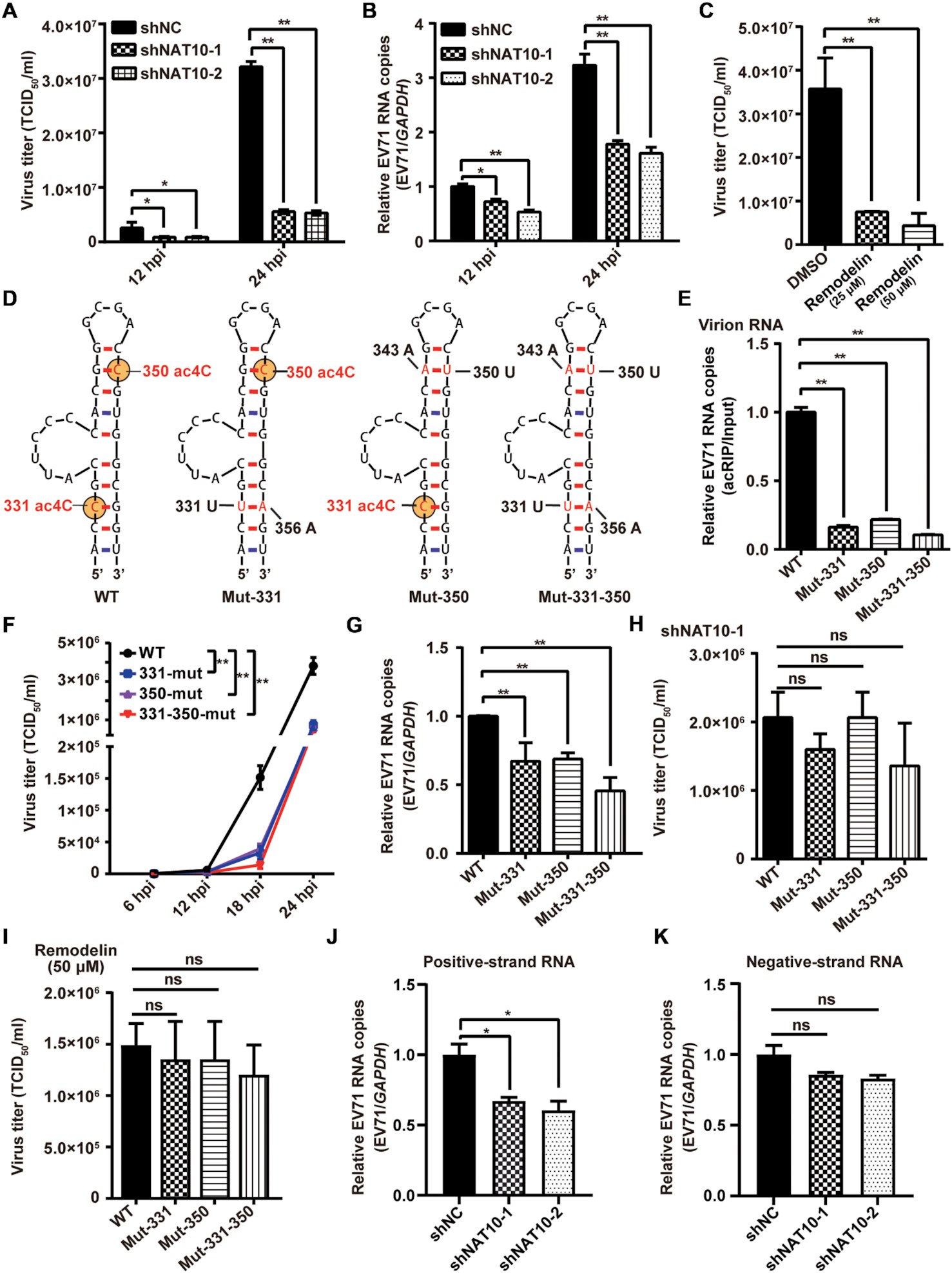
图 2. ac4C 修饰促进了 EV71 的复制
03 ac4C 修饰通过增强 PCBP2 与 IRES 的结合来促进 RNA 的翻译
EV71 RNA 的乙酰化位点位于 IRES 的茎环 IV(nt242-445),对病毒蛋白合成至关重要,因此,ac4C 修饰可能影响 EV71 RNA 的翻译效率。作者用蔗糖密度梯度离心和 qRT-PCR 检测了在有无 NAT10 存在的情况下,核糖体对病毒 RNA 的装载效率。值得注意的是,shRNA 介导的 NAT10 敲除显著抑制了 EV71 RNA 上的核糖体装载,表明 ac4C 增强了 EV71 RNA 的翻译。作者构建了插入 EV71 WT 或 ac4C 突变体的 5』 UTR 区的 eGFP 报告质粒,并将其转染到 RD 细胞中,发现 EV71 ac4C 突变体转染细胞中 eGFP 的表达减少,进一步表明 ac4C 提高了 EV71 RNA 的翻译效率。体外翻译实验同样证实了 ac4C 促进了 EV71 RNA 的翻译。接下来,作者通过 RIP-qPCR 研究了 EV71 WT 或 ac4C 突变体 IRES 与 PCBP2 和 HNRNPK 的结合能力,结果表明,ac4C 通过选择性地增强 PCBP2 与 IRES 的结合来促进病毒的翻译。
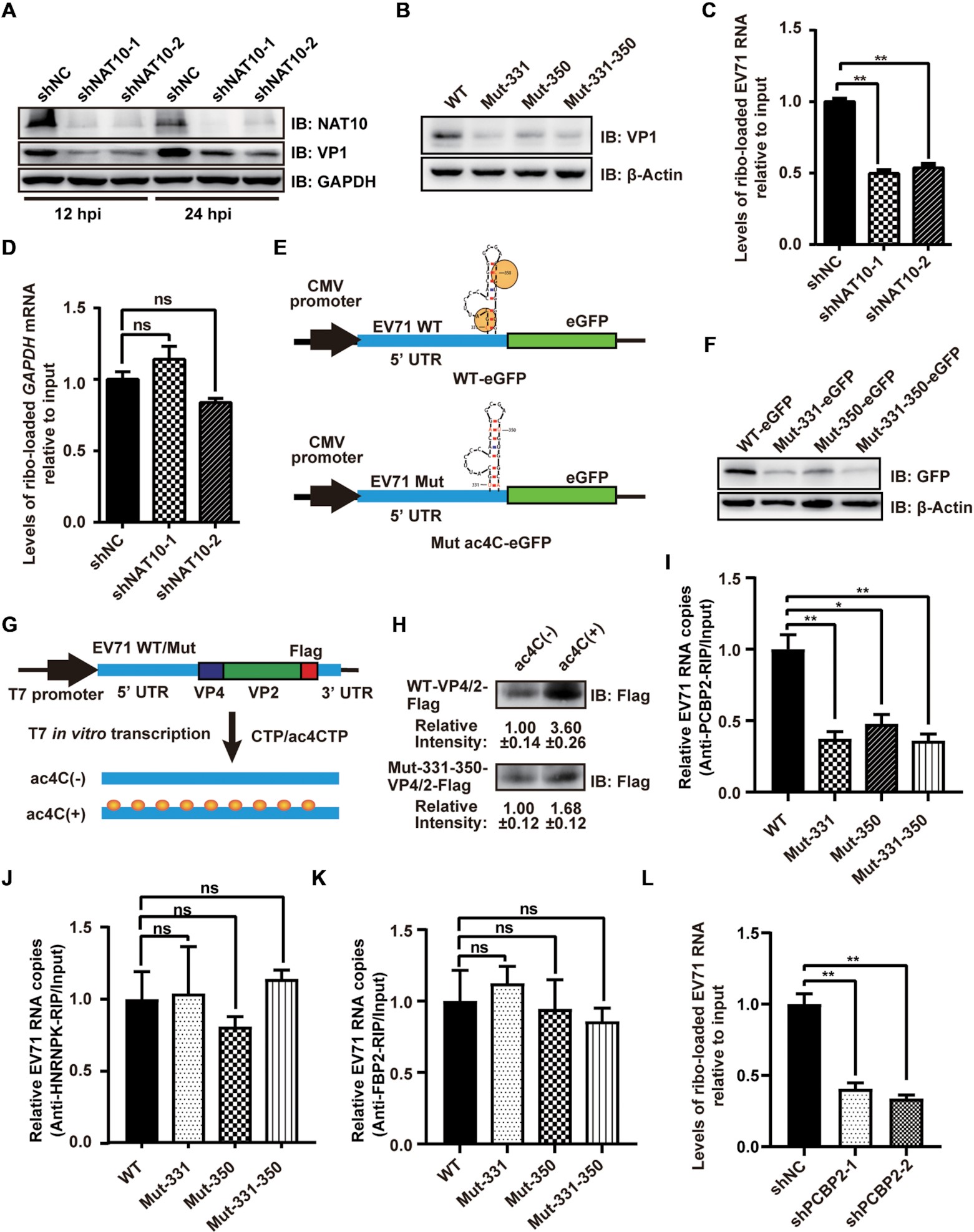
04 ac4C 修饰提高了 EV71 RNA 的稳定性
mRNA 的翻译效率和稳定性有着错综复杂的联系,mRNA 翻译的减少降低了 mRNA 的稳定性,反过来降低了翻译效率。为了探讨 ac4C 修饰是否与 EV71 RNA 的稳定性有关,作者构建了 EV71 WT 和 ac4C 突变体的 3D 移码突变质粒,并将其转染到 Vero 细胞中,在不同时间点进行 qRT-PCR 分析。结果显示,与 WT 病毒相比,ac4C 突变体的 RNA 降解速度加快。然而,当使用重构蛋白抑制 NAT10 的功能时,WT 病毒和突变病毒之间的 RNA 降解率没有显著差异。以上结果表明,ac4C 修饰影响了 RNA 的降解效率。值得注意的是,在 ac4C 修饰的情况下,WT-mut3D 和 Mut-331-350-mut3D 的 RNA 稳定性都增加了,表明 ac4C 增强了 EV71 RNA 的稳定性。接下来,作者探讨了病毒 RNA 翻译对 RNA 稳定性的影响。当使用环 己酰亚 an 处理细胞抑制翻译时,WT-3Dmut RNA 的降解加速,而 ac4C 突变体 RNA 的降解速率没有明显变化。值得注意的是,PCBP2 的敲除导致了病毒 RNA 的加速降解。这些结果表明,ac4C 对 EV71 RNA 稳定性的影响与病毒 RNA 的翻译效率有关。
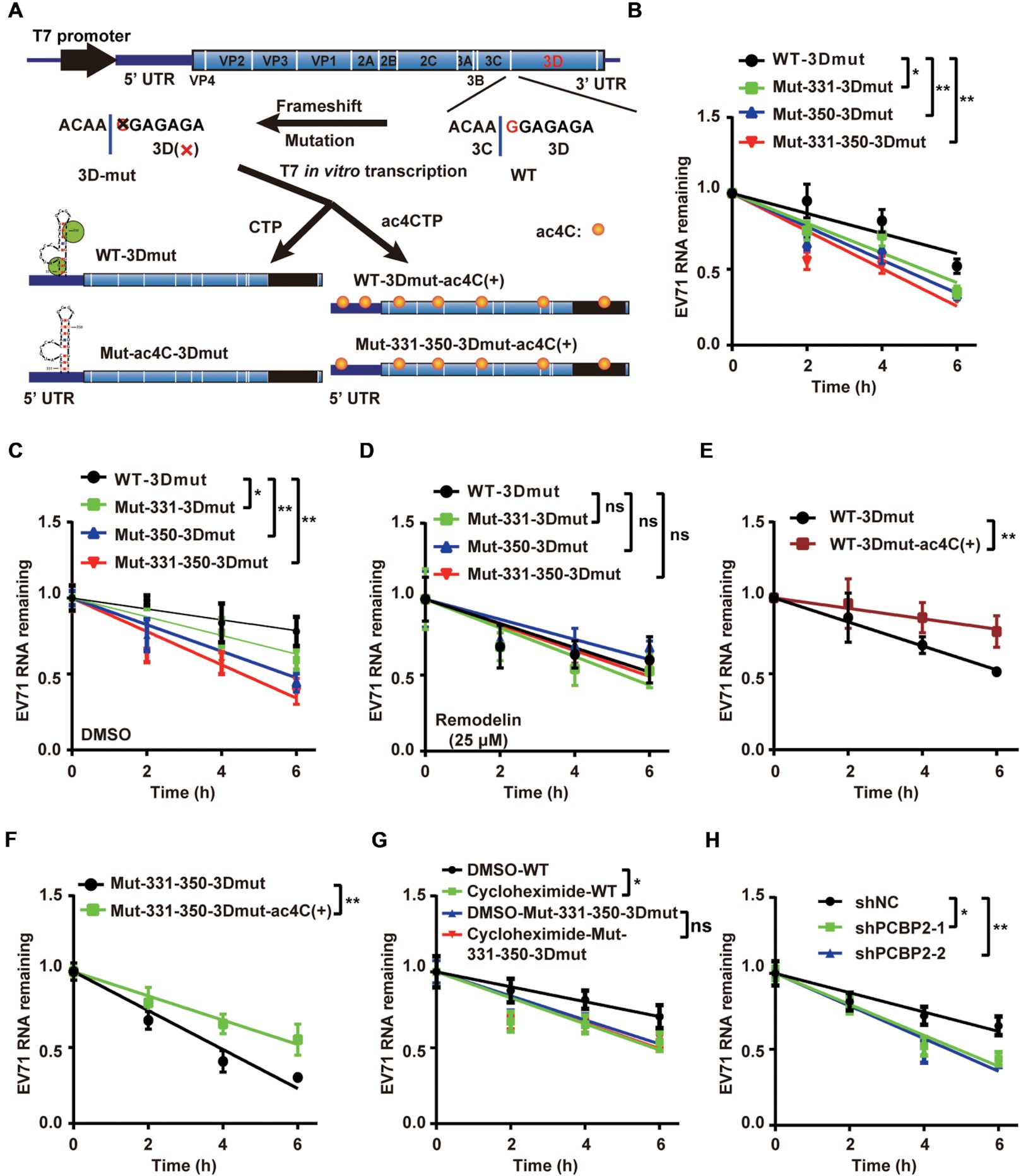
05 ac4C 在体内和体外均促进了 EV71 RNA 与 3D 的结合
3D 是在 EV71 复制过程中直接结合并合成病毒 RNA 的关键蛋白。由于乙酰化促进了 PCBP2 与 EV71 RNA 的结合,作者接下来评估了 ac4C 是否影响了 3D 与病毒 RNA 的结合。作者通过 RIP-qPCR 分析与 3D 结合的 RNA,发现 3D 结合的 EV71 RNA 通过重构蛋白处理抑制 NAT10 而减少,表明 ac4C 修饰影响了病毒 RNA 与 3D 的结合。然而,当 ac4C 突变体病毒与重构蛋白一起培养时,3D 结合的 RNA 并没有明显的变化。这些数据表明,ac4C 修饰促进了 EV71 RNA 与 3D 的结合。为了探索 ac4C 修饰是否影响 3D 与病毒基因组的结合,作者进行了 3D 体外结合试验实验,结果不仅表明 ac4C 在体外增强了 3D 结合,而且表明乙酰化位点的位置对其结合能力具有重要作用。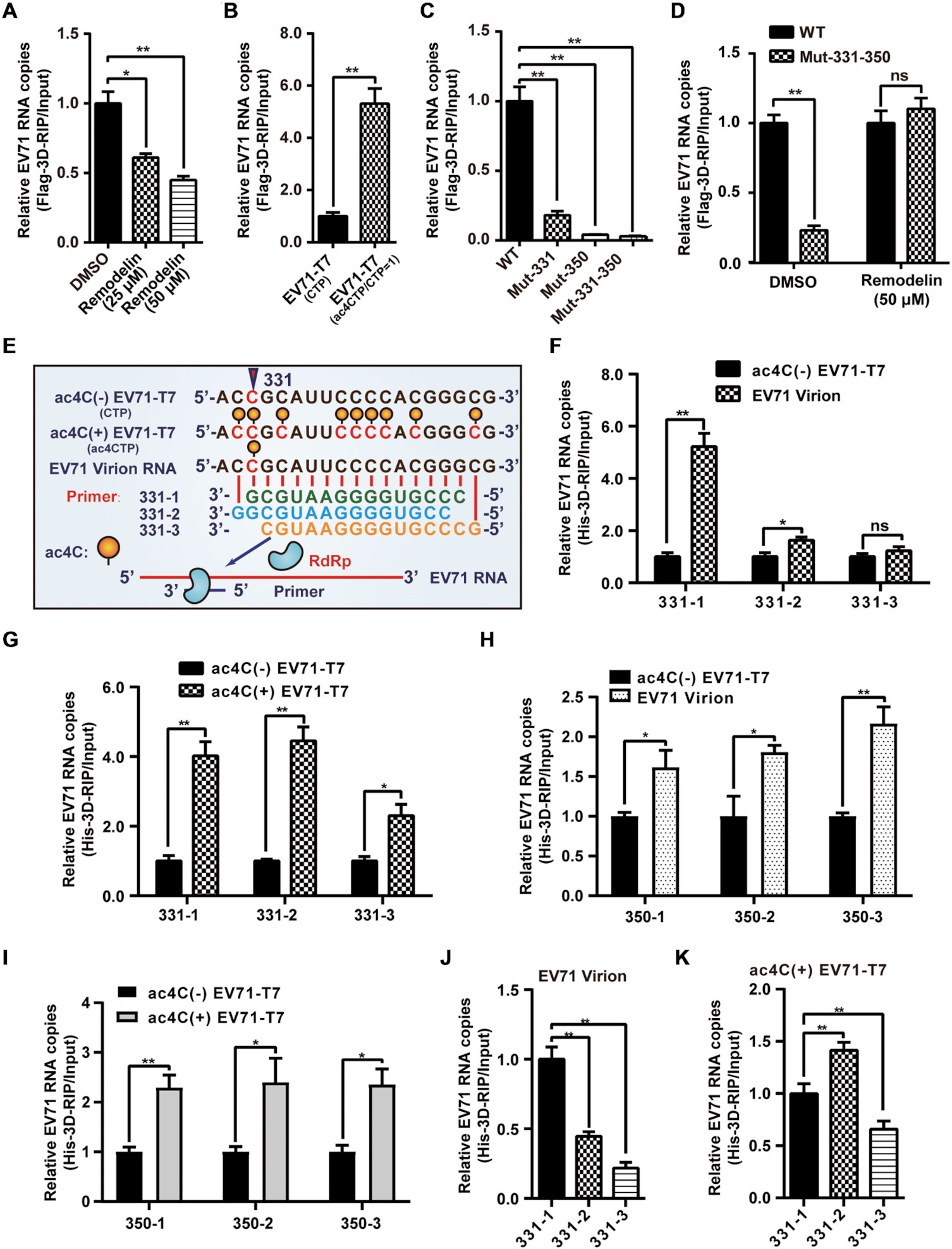
06 EV71 ac4C 突变病毒在小鼠体内的致病性降低
为了研究乙酰化是否影响了病毒的致病性,作者用 EV71 WT 或 Mut-331-350 感染 AG6 干扰素受体缺陷小鼠。与感染 WT 病毒的小鼠相比,感染 ac4C 突变病毒的小鼠显示出更长的存活时间和更慢的体重减轻,且在感染 WT 病毒的小鼠中观察到更严重的肢体瘫痪。此外,qRT-PCR 分析显示,感染 Mut-331-350 病毒的小鼠,所有检测器官的肌肉、肠、脑、心、肝、肝、脾、肺和肾中的病毒 RNA 水平均显著降低。在 mut-331-350 感染小鼠的肢体肌肉、大脑和肠道中观察到致病性降低,导致肌纤维坏死和神经元损伤减少,肠细胞增生、肠绒毛上皮细胞溶解、绒毛顶端细胞空泡化减少。综上所述,这些结果表明 EV71 ac4C 突变减少了小鼠的病理损伤。
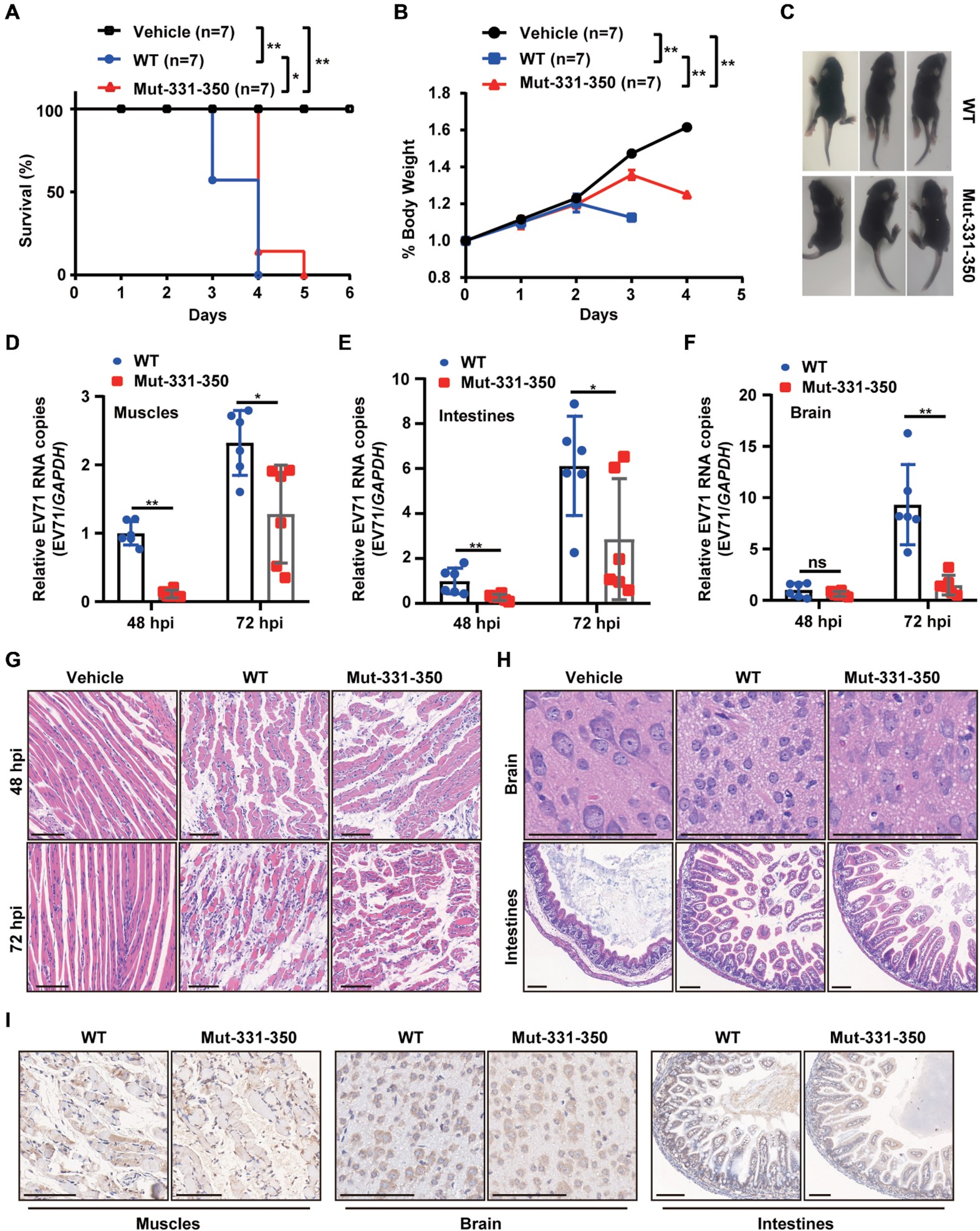
ac4C RNA 修饰测序
对 ac4C RNA乙酰化,目前流行的检测手段为 acRIP-seq 技术,适用于 ac4C RNA 乙酰化谱研究,快速筛选 ac4C RNA 乙酰化靶基因。云序可提供 mRNA 和多种非编码 RNA的ac4C 测序:
- ac4C 全转录组测序(涵盖 mRNA,LncRNA,circRNA)
- ac4C LncRNA 测序(涵盖 LncRNA和mRNA)
- ac4C Pri-miRNA 测序(涵盖 Pri-miRNA和mRNA)
- ac4C mRNA 测序
- ac4C rRNA 测序
- ac4C circRNA 测序
- ac4C tRNA 测序
02 检测整体 ac4C RNA 修饰水平
LC-MS/MS检测整体RNA修饰水平
精准高效,可以实现一次检测,9 类修饰水平检测,一步到位。
03 ac4C RNA 修饰上游酶的筛选
ac4C RNA 修饰相关酶 PCR 芯片
寻找上游直接调控 ac4C RNA 乙酰化的甲基转移酶。
04 ac4C RNA 修饰靶基因验证
acRIP-qPCR
云序提供 acRIP-qPCR 服务,可针对 mRNA,lncRNA,环状 RNA 等不同类型的 RNA 分子进行检测,低通量验证靶基因 RNA 修饰水平。
05 机制互作研究
RIP-seq/qPCR筛选或验证 RNA 修饰直接靶点,研究 RNA 修饰靶基因的调控机制。
RNA pull down -MS/WB
筛选或验证目标 RNA 互作基因或蛋白,研究相应的分子调控机制。
双荧光素酶实验
验证两基因互作,研究相应的分子调控机制。
ChIP-seq
筛选或验证目标蛋白与 DNA 互作,研究相应的分子调控机制。
优势一:云序累计支持客户发表 84 +篇 RNA 修饰 SCI 论文,合计影响因子 814 +,其中 RNA 乙酰化测序高达 3 篇论文 IF 接近 20。
优势二:累计完成数千例 RNA 甲基化测序样本,全面覆盖医口、农口等各类样本。
优势三:全面检测 mRNA 和各类非编码 RNA(circRNA,lncRNA,Pri-miRNA 等)。
优势四:提供ac4C一站式服务:ac4C 整体水平检测、acRIP-seq、acRIP-qPCR 验证、RIP 和 RNA pull down 等。
优势五:率先研发超微量 acRIP 测序技术,RNA 量低至 500ng 起。
优势六:国内最全的 RNA 修饰测序平台,提供 m6A、m5C、m1A、m7G、m3C、m6Am、O8G、ac4C 乙酰化和 2'-O-甲基化测序。
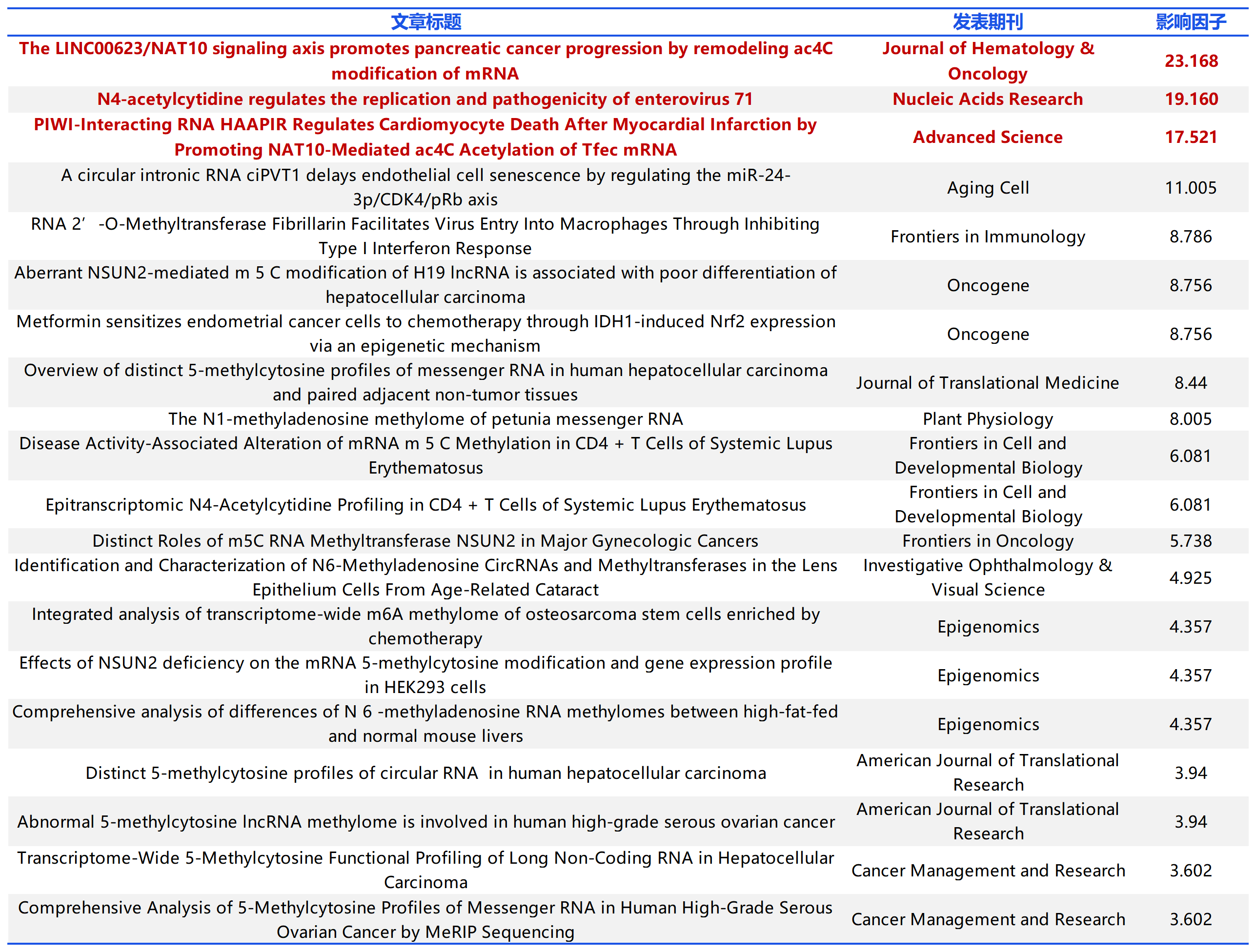
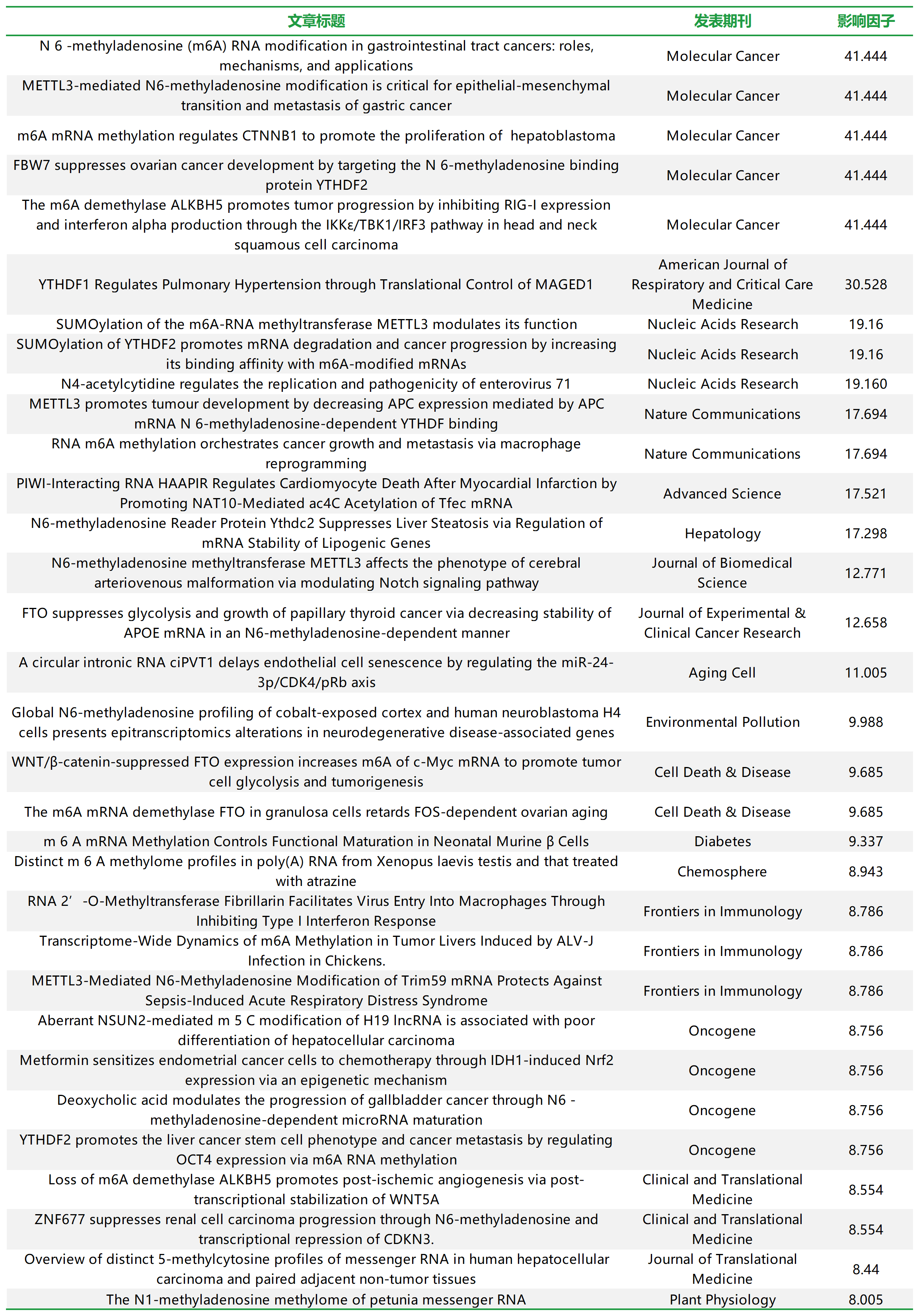
云序客户多种 RNA 修饰(m6A 以外修饰)部分文章列表
相关产品
m5C RNA 甲基化测序
m1A RNA 甲基化测序
m7 G RNA 甲基化测序
ac4C RNA 乙酰化测序
O8 G RNA 氧化修饰测序
2』-O-RNA 甲基化测序
m6Am RNA 甲基化测序
LC-MS 检测整体 ac4C 甲基化水平
RNA 修饰相关酶 PCR 芯片
往期回顾
1 区,IF = 16.8| 云序 RNA 乙酰化测序/acRIP-seq 助力心梗机制研究!
客户文章 1 区 IF 12+ |m6A 甲基化测序助力 FTO 抑制 PTC 糖酵解代谢机制研究
客户文章|新型 RNA 修饰之 m7 G 揭示急性髓系白血病发病机制
GEO 数据上传指南
Nature 新发现:线粒体 m5C 修饰竟是肿瘤转移的元凶!
1 区,IF = 27| 云序 m6A MeRIP-seq 助力鳞状细胞癌机制研究!
1 区,IF = 16.8| 云序 RNA 乙酰化测序/acRIP-seq 助力心梗机制研究!
RNA 甲基化之 m7 G—另辟蹊径带你轻松发文!
超详细图文版:如何实现 m6A 测序数据可视化?
地址: 松江区莘砖公路518号18号2楼
网址: http://www.cloud-seq.com.cn
电话: 021-64878766
邮箱: market@cloud-seq.com.cn




