Cut&tag:新型超微量ChIP-seq技术
发布时间:2020-01-12 15:47 | 点击次数:
文章导读
染色质免疫共沉淀(Chromatin Immunoprecipitation , ChIP)是研究蛋白质与DNA互作的标准方法。通过与高通量测序技术相结合,研究者开发了ChIP-seq技术,从而获得全基因组范围内与组蛋白、转录因子等互作的DNA片段信息,目前被广泛应用于转录调控、表观遗传等研究领域。但是ChIP-seq的应用存在诸多限制:整个过程十分复杂,实验重复性差,需要甲醛交联,并且高度依赖抗体;此外,为了获得有效富集信号,需要大量细胞投入,这使得ChIP-Seq在少量细胞如早期胚胎细胞、干细胞等研究领域力不从心。
新方法、新技术的革命往往给科学研究带来天翻地覆的变化。近期出现的Cut&Tag将繁复的ChIP-Seq变成便捷简单的操作,为研究DNA-蛋白质相互作用提供了有效的工具。2019年4月,弗雷德哈钦森癌症研究中心的Henikoff博士在国际知名学术期刊Nature Communications上发表了Cut&Tag技术,与传统ChIP-Seq方法相比,Cut&Tag技术方法简便易行,信噪比高,重复性好,需要的细胞数量少至60个,且有望将ChIP-Seq做到单细胞水平,开创了ChIP-seq新革命。小编带领大家一起来领略这项前沿技术的风采。

原文链接:Cut&Tag for efficient epigenomic profiling of small samples and single cells
发表期刊:Nature Communications
影响因子:11.878
发表日期:20190429
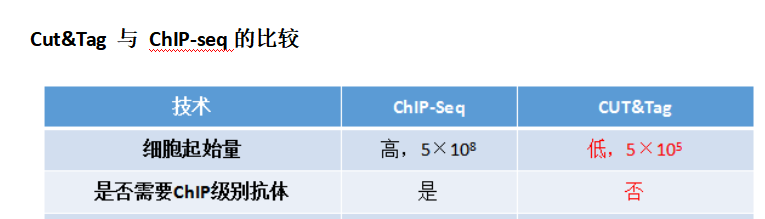
1.Cut&Tag原理
ChiTag是Protein A蛋白与Tn5转座酶的融合蛋白,当抗体结合目的蛋白(如转录因子、组蛋白等)后,Protein A蛋白可直接结合抗体,携带的Tn5转座酶可特异性的切割目的蛋白附近的DNA片段,并将DNA片段连上接头,文库构建后进行高通量测序。
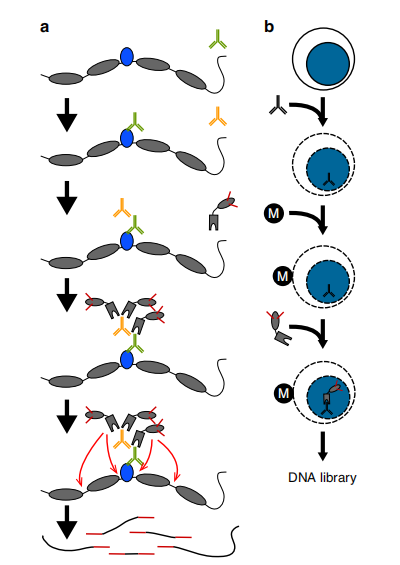
图1 Cut&Tag实验流程图
2. Cut&Tag优势
为证明Cut&Tag技术的有效性,作者在对组蛋白H3K27me3进行研究时对比使用了ChIP-seq、Cut&RUN、Cut&Tag三种方法。从不同角度对三组数据进行比较后,发现Cut&Tag技术具有显著优势,具体结果如下:
(1)背景噪音低
为了直接比较这三种技术,作者将三种方法的数据量都设置为8M Reads,结果发现三种方法得出的峰图相似,但是ChIP-seq的背景噪声非常高,增加数据量到50M Reads时,才能达到类似的峰图,因此ChIP-seq需要更大的测序深度才能将染色质特征与背景区分开。相反,Cut&RUN和Cut&Tag均具有极低的背景噪声水平,其中Cut&Tag的背景噪音最低。

图2 三种方法背景噪音比较
(2)信号值高,灵敏度好
为了量化三种方法的敏感性,作者分别检测了三种方法不同测序深度下峰的富集效率图,发现Cut&Tag可以更快地填充低测序深度的峰,其中约2M Reads相当于Cut&RUN的8M Reads或ChIP-seq的20M Reads,结果表明相同测序深度下,Cut&Tag检测的信号值更高。同时,作者又分析了相同测序深度下(8M Reads),峰图形成的灵敏度,由于ChIP-seq的灵敏度相对较低,所以作者增加了一个40M Reads数据量下的峰图,比较后发现仍旧是Cut&Tag峰图最显著,且比ChIP-seq的40M Reads峰图都高,表明Cut&Tag的灵敏度最高。
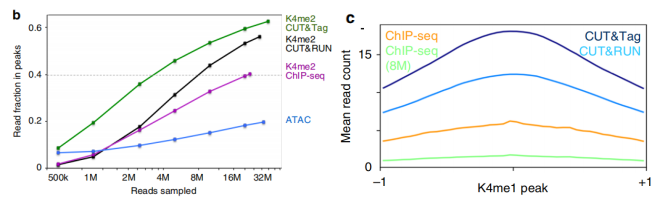
图3 三种方法信号强度比较
(3)重复性好
为了检测三种方法的可重复性,作者分别对三种方法做了两次重复实验,并对实验结果进行分层聚类相关矩阵分析,证明了Cut&Tag实验可重复性最好。
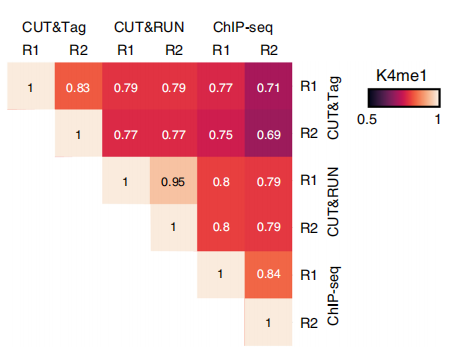
图4 三种方法重复性相关矩阵
总结
Cut&Tag是蛋白质-DNA互作关系研究的新方法,相较于传统的ChIP-Seq具有以下优点:所需细胞量少,背景信号低,可重复性好,无需ChIP级别抗体等优点,甚至可用于单细胞水平测序。Cut&Tag技术可从基因组范围内检测组蛋白、RNA polymerase II和转录因子等蛋白质结合的DNA区端,应用于临床和科研的表观遗传学研究,或是结合生物信息学分析,找到转录因子下游调控的靶基因,为进一步阐明生物学机制提供依据。
云序生物Cut&Tag技术服务:
Cut&Tag(Cleavage Under Targets and Tagmentation)是蛋白质-DNA互作关系研究的新方法,是新一代超微量ChIP-Seq技术,适用于无ChIP级别抗体的蛋白研究。与传统的ChIP-Seq研究方法相比,该技术无需交联、超声打断、末端抹平和接头连接等操作,因此具有省时高效、所需样品量少、背景信号低和可重复性好等优点,甚至可用于单细胞水平测序。Cut&Tag有望将蛋白质-DNA互作的研究变成一种类似PCR反应的常规操作,对基因调控、表观遗传等领域的研究具有革命性的意义。
产品优势
-
样品起始量低:能够对及少量样品进行分析
-
无需ChIP级别抗体:无需提供ChIP级别抗体
-
性价比高:背景噪音低,所需测序深度少
-
实验重复性好:实验重复结果一致性好
实验流程
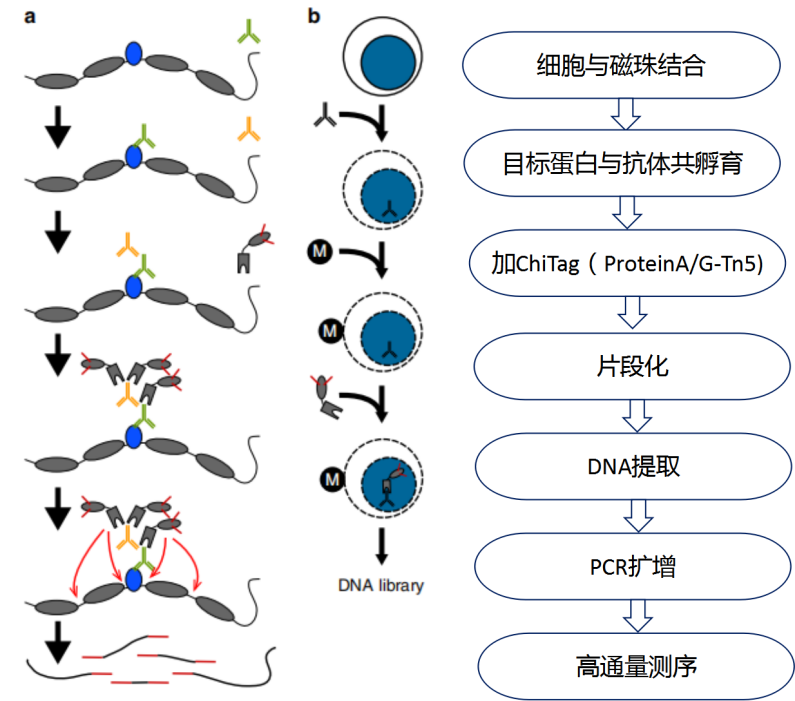
云序生物Cut&Tag实验流程图
样本要求
1)样品类型:细胞、组织,其它样品信息请详询
2)样品量
细胞:5×105
组织:5mg
3)样品保存:细胞样品或新鲜组织块,液氮冻存后,-80℃ 保存。
4)样品运输:干冰运输。
云序生物分子互作技术:
云序作为一家依托于高通量测序的科研服务公司,除了提供优质测序服务,还为客户提供机制研究的服务,助力用户发表高分文章,其中包括研究蛋白互作的CoIP技术、RNA与蛋白/RNA互作的RNA pull-down技术、蛋白与DNA互作的ChIP和ATAC技术,近期更推出了新一代ChIP-seq技术Cut&Tag,囊括了DNA、RNA和蛋白质这三类经典大分子互作研究的重要技术。
云序生物,您身边的测序专家!
云序生物相关产品推荐
云序生物往期回顾
Cebola Inês,Pancreatic Islet Transcriptional Enhancers and Diabetes.[J] .Curr. Diab. Rep., 2019, 19: 145.
上海云序生物科技有限公司
Shanghai Cloud-seq Biotech Co., Ltd.
地址:上海市松江区莘砖公路 518 号 20 号楼 3 楼
电话:021-64878766
传真:021-64878766
网址:www.cloud-seq.com.cn
邮箱:market@cloud-seq.com.cn




