新型RNA修饰m1A&m7G齐登nature 子刊
发布时间:2020-01-12 10:12 | 点击次数:
“表观转录组学”是通过转录后修饰影响RNA的结构和功能,是近几年来生物学科里最热门的研究领域之一,目前已知的RNA修饰类型超过150种,其中包括m6A、m5C、m1A、m7G、2’-氧-甲基化、ac4C RNA乙酰化等。近期小编发现在探究RNA修饰领域中,各位m6A研究领域的鼻祖们又有了新的动作。今年9月份,何川教授团队在Nature Methods发布了m1A甲基化修饰,中国科学院北京基因组研究所杨运桂及杨莹教授在Cell Research上发布了m7G甲基化修饰。继m6A成为明星之后,m5C、m1A、m7G、2’-氧-甲基化、ac4C RNA乙酰化这些明日之星也逐渐彰显潜力。
何川教授助力m1A甲基化修饰研究
2019年9月23日,美国芝加哥大学Bryan C. Dickinson团队与何川团队在Nature Methods上发表“Evolution of a reverse transcriptase to map N1-methyladenosine in humanmessenger RNA”研究成果,该项研究开发并验证了一个逆转录酶进化平台,可快速选择逆转录酶,使其在逆转录过程中越过RNA修饰位点,并使其发生突变,从而特异性识别RNA修饰。该平台首先被应用到了对m1A修饰的检测,在测序过程中产生突变标记,最终实现单碱基识别m1A,为m1A生物学的研究提供了新的工具、分析方法和数据集。
发表期刊:Nature Methods
影响因子:28.467
实验方法:m1A-seq
全文链接:https://sci-hub.se/10.1038/s41592-019-0550-4
研究内容
作者将筛选的HIV病毒逆转录酶应用于m1A-seq中,并分别在rRNA和tRNA中检测了m1A的突变特征。结果显示,在加入AlkB处理后,28 S rRNA 中m1A1322的突变率分别由78%和67%,下降到了21%和5%。作者观察了38个细胞质tRNA序列中m1A58的突变特征,发现AlkB处理后,也表现出了约86%突变率降低。同时,作者也检测了线粒体mRNA ND5 (chrM:13711)、PRUNE mRNA (chr1: 150980982)、lncRNA MALAT1 (chr11: 65273630)和28S rRNA中A1322位点的m1A突变率,这些结果都显示,加入AlkB处理后,高于50%的m1A突变率的下降。这说明,利用HIV病毒逆转录酶应用于二代测序能够量化和比较不同生物环境下各分子中的m1A修饰情况。
杨运桂/杨莹教授助力m7G表达谱研究
m7G是tRNA、rRNA和mRNA 5’帽子种最丰富的修饰之一,在调节RNA加工、代谢和功能方面具有重要作用。目前研究发现,m7G除了存在于mRNA的帽子区,还存在于mRNA内部区域。然而,其在转录组范围的分布和mRNA内部的调控仍然未知。
鉴于此,2019年9月13日,中国科学院北京基因组研究所杨运桂及杨莹教授在Cell Research上发表题目"Dynamic methylome of internal mRNA N7-methylguanosine and its regulatory role in translation" 的研究成果,该研究利用m7G-seq特异性检测mRNA内部m7G修饰。并且,揭示了m7G能够显著富集在mRNA 5’UTR区域和AG富集,在不同的人/小鼠细胞系及组织中具有良好的特征。值得注意的是,mRNA内部m7G修饰在H2O2和热休克处理下能够被动态调控,并且在CDS和3’UTR区域显著被富集,在促进mRNA翻译效率方面发挥作用。
该研究揭示了mRNA 内部m7G甲基化修饰的动态特征,并突出显示m7G可以考虑作为一种新的表观转录组标记。

发表期刊:Cell Research
影响因子:17.848
实验方法:m7G-seq
实验对象:人类细胞系(Hela、293T)、小鼠胚胎干细胞、小鼠脑组织
全文链接:https://sci-hub.se/10.1038/s41422-019-0230-z
研究内容
(1)mRNA内部m7G的基因组特征
该研究,作者通过m7G-seq方法检测了人类细胞系(Hela、293T)中m7G的分布情况,结果显示m7G主要富集在mRNA的起始密码子区,分布具有细胞系中具有很好的保守性。且无论m7G发生在5’ UTR、3’ UTR区,还是CDS区均能提高mRNA的翻译效率。
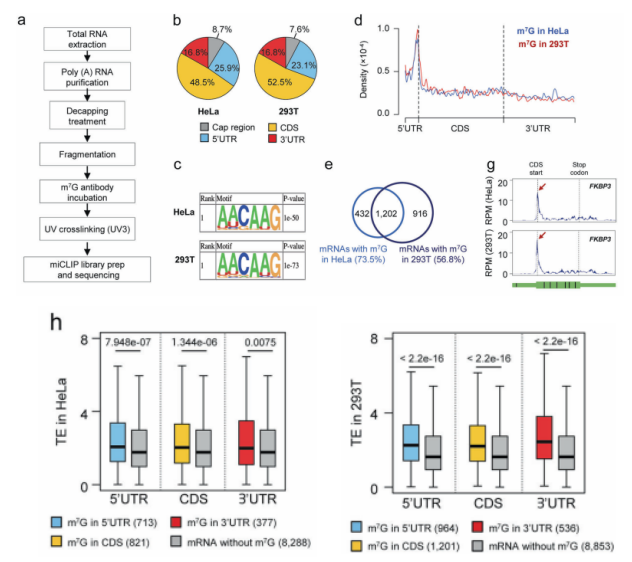
(2)mRNA内部m7G具有物种保守性
有研究报道,m7G在原核生物,真核生物和古菌中都是保守的。因此作者对小鼠胚胎干细胞(mESC)和小鼠脑组织进行m7G-seq,发现小鼠中m7G基因组特征分布情况和人的具有高度一致性。

(3)H2O2和热休克能够动态调控mRNA内部m7G修饰
有研究发现,m6A和m5C在氧化应激诱导的细胞衰老过程中能够协同增强p21的表达,同时m6A能够参与热休克反应中HSP表达的调节,因此作者进一步探讨mRNA内部m7G是否也可以在压力耐受条件下作为一种新表观调控因子调控mRNA的代谢。因此作者对H2O2和热休克处理人293T细胞,进行m7G-seq,引人注目的是,H2O2和热休克处理下能够动态调控mRNA 内部m7G修饰,并且在CDS和3’UTR区域显著被富集,同时其在促进mRNA翻译效率方面也发挥作用。

H2O2和热休克处理下能够动态调控mRNA内部m7G修饰
内部m7G促进PCNA mRNA翻译
总结
小编为大家解析的这两篇高分文章中,何川教授和杨运桂/杨莹教授都开始研究m1A和m7G甲基化修饰,进一步丰富了m1A和m7G的研究价值和意义。随着科研工作者们对RNA修饰研究的不断探索,各类RNA修饰的功能和机制也不断被揭示,不仅研究比较火热的m6A、m5C,还有一些新修饰如m1A、m7G、ac4C、2’-氧-甲基化等,这对于揭示并探索表观转录组学新视角具有非常重要的意义。但是,目前对于RNA修饰的研究仍处于起步阶段,并且其在各种疾病和领域中具有非常重要的研究价值和潜力。而云序生物作为较早提供RNA修饰服务的公司,在行业中起到了一定的模范和领导作用,优秀的研发团队也使得公司在行业中具有非常强的竞争力。
云序生物RNA表观转录组学特色:
云序作为一家依托于高通量测序的表观转录组学科研服务公司,长期致力于提供优质前沿表观研究测序产品,继m6A、m5C、m1A之后,云序隆重推出多款RNA新修饰类型,不仅有ac4C RNA乙酰化测序,还包括m7G/m3C RNA甲基化测序、2'-氧-甲基化测序等产品,打造了全面的RNA修饰研究平台。
云序,您身边的表观转录测序专家!
云序生物相关产品推荐
RNA pull down 测序
云序生物往期回顾
“雨露均沾”---- RNA甲基化“搭讪”成熟miRNA又出新花样
m6A甲基化赶迟了怎么办?m5C甲基化正在风口上
云序生物2019国自然解读!
云序客户教你如何快速发表5分的m6A甲基化文章
Cell热点: RNA乙酰化——解密m6A热点之后RNA修饰新方向
胖纸的福音!敲除Zfp217基因可通过RNA m6A带走你腰间的“救生圈”
占领C位!云序生物解析如何做到快速同时检测各类癌症当中RNA甲基化相关酶&RNA甲基化水平(上)
占领C位!云序生物解析如何做到快速同时检测各类癌症当中RNA甲基化相关酶&RNA甲基化(下)
云序客户12分顶级文章,教你如何巧用RIP测序玩转分子机制!
Nat. commun | Mettl3介导的m6A RNA甲基化调控骨髓间充质干细胞和骨质疏松症的命运
Nature | SMAD2/3与TGF-β通路协同影响转录因子发生m6A RNA甲基化调控干细胞发育
Nature | m6A RNA甲基化识别蛋白YTHDF1参与记忆的形成
上海云序生物科技有限公司
Shanghai Cloud-seq Biotech Co., Ltd.
地址:上海市松江区莘砖公路 518 号 20 号楼 3 楼
电话:021-64878766
传真:021-64878766
网址:www.cloud-seq.com.cn
邮箱:market@cloud-seq.com.cn




